Le BioMedical Model Hub (BimmoH) est la dernière initiative de l’EURL ECVAM (au sein du Joint Research Center – JRC), lancée en 2025, pour appréhender l’explosion des modèles non animaux émergeant de la recherche biomédicale. Bien plus qu’une simple base de données, utilisant l’intelligence artificielle, BimmoH est conçu pour extraire, filtrer et catégoriser les publications scientifiques (sur Pubmed) qui utilisent des modèles basés sur la biologie humaine permettant l’utilisation d’approches non animales.
BimmoH, coordonnée par le JRC et la société Fresci, est la plus grande base de données publique d’articles scientifiques utilisant des modèles de biologie humaine en recherche biomédicale, continuellement mise à jour.
Elle doit permettre, entre autres applications, d’actualiser et se substituer aux revues de l’ECVAM des méthodes non-animales par domaine pathologique.
Les objectifs et méthodes de BimmoH sont précisés dans cette présentation du 12 novembre 2025.
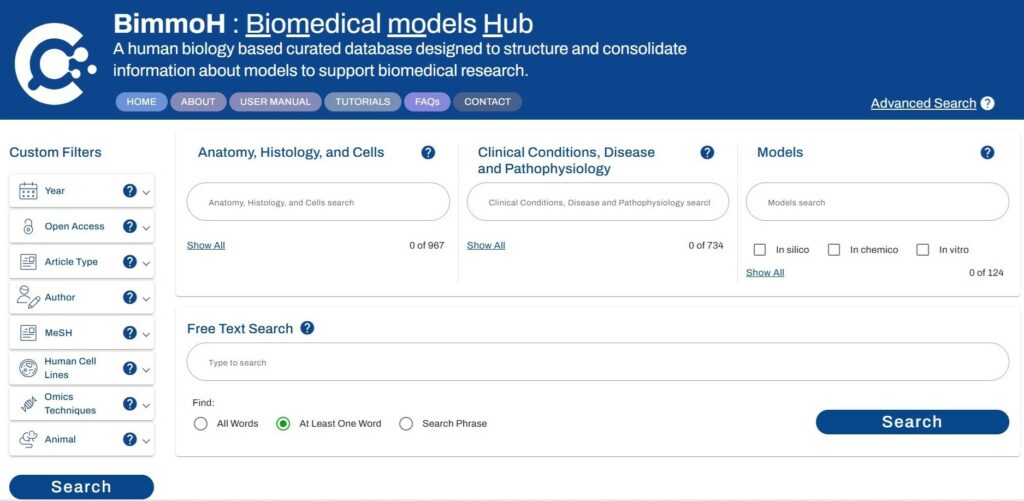
L’IA trie les modèles selon trois grandes catégories :
- Anatomie, histologie et types de cellules (p. ex. foie, cerveau, peau)
- Situations cliniques et domaines pathologiques (p. ex., cancer, maladies respiratoires)
- Type de modèle (modèles in silico, in vitro (organoïdes, organes sur puce…), in chemico)
Il est possible aussi de lancer une recherche en texte libre. Toutefois, pour éviter d’être noyé sous des centaines de publications, mieux vaut utiliser aussi tout ou partie des trois catégories ci-dessus et/ou des filtres figurant sur la colonne de gauche : années, type d’article, lignées cellulaires, etc.
Une requête permet d’obtenir une liste de publications pertinentes très rapidement.
Page d’accueil :
Lire sur le site de ProAnima l’interview de Marco Straccia, neuroscientifique et fondateur de l’agence de conseil FRESCI, qui a livré BimmoH (décembre 2025).
Cette base présente quelques limites :
- Elle s’appuie uniquement sur la base Pubmed, qui certes est la principale base en matière de publications dans le domaine biomédical (et gratuite), mais il existe d’autres bases de données, comme « web of science ».
- De nombreuses publications proposées mêlent des méthodes sans animaux et des tests sur animaux, si bien que le remplacement n’est que partiel.
- L’outil ne constitue pas encore un outil permettant d’identifier des pistes de recherche via des méthodes sans animaux sur la base d’une question posée, et à cet égard l’intelligence artificielle n’est pas encore utilisée dans sa pleine capacité, même s’il ne faut pas masquer les difficultés techniques d’une telle évolution (peut-être dans une version ultérieure ?).
- Il existe d’autres outils poursuivant les mêmes objectifs, à savoir identifier des publications dans la base Pubmed ayant mis en oeuvre des méthodes de recherche sans animaux, et aucune coordination n’est faite entre ces outils.
Parmi ces autres outils, on peut citer la base NAT (non animal technologies), développée à l’initiative de l’association allemande « Ärzte gegen Tierversuche », ou aussi Smafira (acronyme pour « SMArt Feature based Interactive RAnking »), développé à l’initiative du BF3R en Allemagne, qui, pour un article de référence, fournit des articles similaires dans le même domaine et propose des articles ayant développé des méthodes alternatives. Ces deux outils travaillent aussi à partir de la base Pubmed.
Quoi qu’il en soit, BimmoH constitue un progrès important dans l’accès à l’information sur les méthodes non-animales et mérite qu’il soit largement utilisé par les chercheurs.
Il devrait impérativement être utilisé de manière systématique par les comités chargés de l’évaluation des projets, pour évaluer les possibilités de remplacement face à une demande d’autorisation de projets.




